
Professeur des universités
manoel.manghi(at)irsamc.ups-tlse.fr
05 61 55 61 77
326

 Mes recherches portent sur des questions qui se situent à la frontière de la physique de la matière molle et de la biophysique. Les techniques utilisées sont variées et vont des approches analytiques utilisant la théorie statistique des champs, les approches variationnelles gaussiennes, les matrices de transfert, les lois d’échelles, les équations hydrodynamiques (avec ou sans bruit) aux simulations numériques de dynamique Brownienne ou Monte Carlo [les numéros se rapportent aux publications].
Mes recherches portent sur des questions qui se situent à la frontière de la physique de la matière molle et de la biophysique. Les techniques utilisées sont variées et vont des approches analytiques utilisant la théorie statistique des champs, les approches variationnelles gaussiennes, les matrices de transfert, les lois d’échelles, les équations hydrodynamiques (avec ou sans bruit) aux simulations numériques de dynamique Brownienne ou Monte Carlo [les numéros se rapportent aux publications].
 domaines lipidiques et/ou protéiques à l’échelle de la cellule [48, 55]
domaines lipidiques et/ou protéiques à l’échelle de la cellule [48, 55]


2010 Habilitation à Diriger des Recherches, Physique statistique d’objets biologiques et des électrolytes aux interfaces, LPT, Univ. P. Sabatier, Toulouse.
depuis 02/2005 Maître de Conférences, LPT, Université Toulouse III -Paul Sabatier
09/2004-01/2005 Contrat postdoctoral CNRS Laboratoire de Physique Théorique et Astroparticules, Montpellier
2002-2004 Contrat postdoctoral dans le groupe du Pr. R.R. Netz, Ludwig Maximilian University, Munich. Bourse de recherche de la fondation allemande Alexander von Humboldt
1999-2002 Thèse de doctorat en physique théorique, Contributions théoriques à l’étude des polymères aux interfaces, Laboratoire Structures et Propriétés des Architectures Macromoléculaires, Université J. Fourier, Grenoble.
1995-1999 Elève de l’École Normale Supérieure de Lyon
1998 Agrégé de sciences physiques, option physique.
Ce Master a pour but de former des étudiants de haut niveau en biophysique, physico-chimie, matière molle, physique de l’imagerie, physique des comportements sociétaux afin qu’ils soient en mesure d’appréhender des problèmes de biologie avec les outils de la physique. Ceux-ci sont maintenant très utilisés en sciences du vivant à la fois pour observer et étudier le vivant (microscopie, analyses de données, suivi de trajectoires…) mais aussi pour modéliser et comprendre les mécanismes physiques à l’origine des phénomènes biologiques. Le Master PFA-PMV aborde les différentes échelles du vivant, de l’échelle moléculaire (ADN, membranes…) à l’échelle des populations (fourmis, poissons, humains…) en passant par l’échelle cellulaire (bactéries…) et des tissus biologiques (épithelium, sang…). Voir le lien suivant.
Le Master 1 se poursuit ensuite en Master 2 Physique pour le Vivant.
Pour plus d’informations voir le site dédié à l’adresse web page
MobiGen Allocation Doctorale Interdisciplinaire Uninversité de Toulouse-Région Occitanie (2022) Dynamique du génome (avec Kerstin Bystricky)


La rigidité de l’ADN double-brin joue un rôle majeur dans la structuration du chromosome et donc dans l’expression des gènes, ainsi que dans les nanotechnologies où l’ADN est utilisé comme élément de construction. Mais comment cette rigidité est-elle influencée par la présence de différents types d’ions ? Dans ce travail, des équipes de l’IPBS et du LPT de Toulouse ont répondu à la fois expérimentalement et théoriquement à cette question.
Grâce à la parallélisation massive de la technique de Tethered Particle Motion (TPM), ils ont mesuré la dépendance de la longueur de persistance, qui reflète la rigidité de l’ADN, sur une large gamme d’ions et de concentrations de sels. Ils ont mis en évidence une décroissance unique pour les ions métalliques monovalents ou divalents, décrite par des théories récentes, qui prennent en compte les effets électrostatiques non linéaires ainsi que le diamètre fini de l’ADN. Cette étude permettra ainsi de prédire les changements de conformation des structures complexes formées par l’ADN tant in vitro qu’in vivo.
Référence: S. Guilbaud, L. Salomé, N. Destainville, M. Manghi, and C. Tardin, Dependence of DNA Persistence Length on Ionic Strength and Ion Type. Physical Review Letters, 122, 028102 (2019).
A.K. Dasanna, N. Destainville, J. Palmeri et M. Manghi du LPT ont étudié théoriquement le mécanisme de fermeture des bulles de dénaturation de l’ADN et ont montré que la courbure des chaînes et la diffusion des brins sont à l’origine des longs temps de fermeture mesurés expérimentalement.
Au cours de la transcription de l’ADN, des bulles de dénaturation (segments d’ADN où des paires de bases sont ouvertes) sont nucléées dans l’ADN et une question importante concerne le temps de fermeture de ces « bulles » d’ADN. Des expériences ont permis de mesurer des temps de fermeture étonnamment longs, de l’ordre de 20 à 100 microsecondes, pour de petites bulles d’une longueur d’environ 20 paires de bases.

En utilisant des simulations de dynamique brownienne et des arguments analytiques, il a été montré que la fermeture d’une bulle pré-équilibrée (a) se produit en deux étapes. La première étape consiste en une fermeture rapide de la bulle initiale jusqu’à ce qu’un état de bulle métastable d’une longueur d’environ 10 paires de bases soit atteint (b). La force motrice de cette cinétique rapide est le gain énergétique dans la fermeture des paires de bases, qui devient interdit à un moment donné par la grande courbure stockée dans la bulle. La fermeture de cet état métastable est alors contrôlée par la diffusion rotationnelle des deux bras rigides (c). Pour les ADN réels, le temps de fermeture est égal à N^2.4 pour des longueurs d’ADN N comprises entre 20 et 100.
Référence : A.K. Dasanna, N. Destainville, J. Palmeri and M. Manghi, Strand diffusion-limited closure of denaturation bubbles in DNA, Europhysics Letters 98, 38002 (2012)
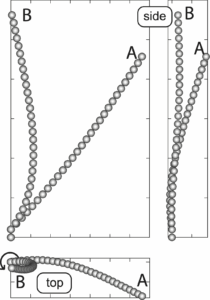
La microscopie à force atomique (AFM) est largement utilisée pour observer l’ADN double-brin adsorbé sur des surfaces. Dans des expériences menées par Wiggins et al., des « anomalies » ont été détectées dans la distribution des angles de courbure le long de l’ADN (qui mesure sa flexibilité) : une surabondance de grands angles a été trouvée, qui n’est pas prédite par le modèle statistique traditionnel de l’ADN, le modèle de ver (voir figure). N. Destainville, M. Manghi et J. Palmeri du LPT ont expliqué ces anomalies par la présence de petites bulles de dénaturation (ou kinks) facilitées par la présence du substrat qui modifie les interactions entre les paires de bases de l’ADN. Ils prédisent que ces anomalies existent en 3D mais qu’elles sont trop faibles pour être détectées et qu’elles réconcilient la discordance apparente entre les propriétés élastiques observées en 2D et en 3D. Par conséquent, les conclusions sur les propriétés 3D de l’ADN (et des protéines et enzymes qui l’accompagnent) ne découlent pas directement des expériences 2D réalisées par AFM.
Référence : N. Destainville, M. Manghi et J. Palmeri, Microscopic mechanism for experimentally observed anomalous elasticity of DNA in 2D, Biophysical Journal 96, 4464 (2009). abstractarxiv0903.1826
La dénaturation de l’ADN est un processus physique au cours duquel la double hélice peut s’ouvrir localement grâce à des fluctuations thermiques. L’ouverture de paires de bases successives crée une bulle de dénaturation. À l’intérieur d’une bulle, les deux brins simples fluctuants ont une rigidité de flexion 50 fois plus faible que celle de l’hélice non ouverte. Il s’ensuit qu’à une température donnée, l’ADN peut explorer un nombre beaucoup plus important de configurations géométriques lorsqu’il est à l’état de bulle et donc augmenter son entropie conformationnelle. De cette manière, il peut également présenter une courbure locale plus importante, par exemple lorsqu’il est enroulé autour d’un histone. La géométrie externe de l’ADN influencera à son tour le processus de création de bulles. Cette influence mutuelle conduit naturellement à un modèle théorique couplant les états internes locaux de l’ADN (paires de bases ouvertes ou fermées) et la courbure locale de l’ADN.

Référence : J. Palmeri, M. Manghi, and N. Destainville, Thermal Denaturation of Fluctuating DNA Driven by Bending Entropy, Physical Review Letters 99, 088103 (2007).
En couplant les propriétés élastiques d’un nano-filament semi-flexible rotatif et les interactions hydrodynamiques, à l’origine de la propulsion dans les écoulements de Stokes, la propulsion bactérienne peut être reproduite de manière biomimétique. Ces travaux mettent en lumière le rôle majeur joué par l’élasticité dans le mouvement flagellaire de bactéries comme E. coli.Les flagelles bactériens sont des polymères rigides hélicoïdaux mis en mouvement à leur base par un moteur rotatif. En utilisant des simulations de dynamique brownienne avec des interactions hydrodynamiques complètes qui prennent en compte la diffusion de la quantité de mouvement dans l’écoulement de Stokes, il a été montré qu’un simple nano-filament élastique rigide mis en rotation subit, à un couple critique, une bifurcation de forme fortement discontinue vers un état hélicoïdal. Il produit ainsi une poussée substantielle vers l’avant, quel que soit son sens de rotation. Par conséquent, dans un contexte biomimétique, il rend inutile l’utilisation d’un polymère hélicoïdal, les polymères rigides droits étant beaucoup plus simples et abondants. Enfin, ces effets élastiques pourraient expliquer certaines observations faites sur E. coli, telles que les transformations polymorphes de leurs flagelles.
Référence : M. Manghi, X. Schlagberger, R.R. Netz, Propulsion with a Rotating Elastic Nanorod, Physical Review Letters, 96068101 (2006)